清华学者利用 CRISPR 敲低技术一次性研究细菌中数千个基因
6 月 26 日,清华大学化工系生物育种技术与装备团队在《自然·通讯》(Nature Communications)发表 文章,报道了一种基于 CRISPR/dCas9 敲低技术 (CRISPRi) 的新型细菌功能基因组学方法,可以一次性研究细菌中数千个基因和特定表型的关系。
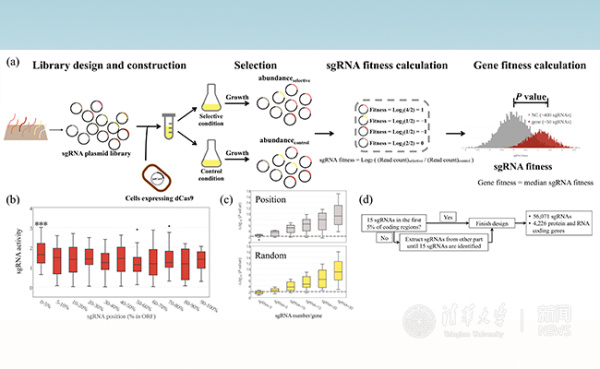
经过工程化改造的细菌免疫 CRISPR 系统(Clustered regularly interspaced short palindromic repeats)已经发展成为具有广泛应用的基因组编辑工具。近年来,科学家逐渐展示了其在高通量基因功能筛选方面的应用潜力:首先对待筛编码基因进行敲除或敲低,并以细胞生长或特定标记作为一个表型指标,将靶定待研究表型相关基因的单导向 RNA(sgRNA)文库富集出来,并利用高通量测序技术定量每个基因的功能。目前这一策略主要被用来进行高等真核生物的高通量功能基因组学研究。由于细菌和高等真核生物基因组的系统性结构差异,在细菌中使用这种方法的高通量功能筛选尚未见报道。在这项最新研究工作中,清华大学研究团队构建了大肠杆菌全基因组范围的 sgRNA 文库,结合 CRISPRi 基因敲低技术,对于这一模式微生物的一系列重要基因组学问题开展了大规模研究。
利用合成 sgRNA 文库和 CRISPRi 基因敲低技术进行高通量细菌功能基因组学研究
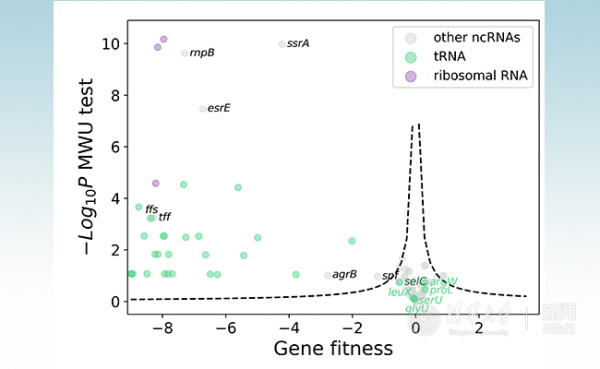
研究团队首先证明了这一方法可以有效地鉴定出大肠杆菌的已知必需基因,同时也对必需基因筛选结果中和已知数据库不吻合的部分基因进行了进一步实验验证,更新了对于该模式微生物必需基因的认识。此外,他们还发现该方法可以有效地研究细菌中 RNA 编码基因的功能。尽管这类基因近年来被发现广泛的参与细菌的重要生物学过程,但是由于传统细菌高通量遗传分析方法本身的局限,这类基因受到了普遍的忽视。有趣的是,利用该方法绘制的转运 RNA(tRNA)必需性图谱,研究团队发现了大肠杆菌中有六个非必需的 tRNA 编码基因,且不同于 tRNA 普遍的多拷贝特性,这六个 tRNA 编码基因都以单拷贝形式存在于大肠杆菌基因组中。
新型 CRISPRi 混合筛选鉴定细菌 RNA 编码基因功能
利用随机转座子插入文库的混合筛查(Tn-seq)是目前应用最为广泛的高通量细菌系统遗传分析手段。研究团队以已知必需基因为标准,系统分析了新建立的 CRISPRi 敲低筛选方法相比于转座子测序的性能优劣。结果表明,在类似文库规模条件下,CRISPRi 敲低筛选方法相比于随机转座子插入文库的混合筛查方法,能够以更低的假阳性率捕获更多的已知必需基因,且编码区域长度越短这一优势越为明显。
研究团队还以氨基酸代谢为例,通过同工酶功能、色氨酸途径环境响应等案例展示了本研究建立的 CRISPRi 方法定量解析微生物代谢网络结构的能力。同时,通过对于大肠杆菌全基因组范围内对于糠醛和异丁醇耐受性图谱的定量分析,发现了诸多已知和未知的耐受性基因位点,为菌株的工程化改造提供了基础。
该工作第一次报道了利用 CRISPRi 方法结合高通量混合筛选和测序对于细菌的功能基因组学进行研究,并通过对于结果的系统分析提出了适用于细菌基因组结构的 sgRNA 文库设计原则。值得一提的是,为了方便广大研究者使用这一方法,研究团队还开发了软件工具,可以一站式的进行 sgRNA 文库的设计和筛选后的测序数据分析。
化工系张翀副教授为本文通讯作者,生物育种技术与装备团队首席邢新会教授,信息国家研究中心、自动化系谢震研究员为本文共同作者,他们同时为清华大学合成与系统生物学研究中心核心成员。清华大学化工系博士生王天民、关长阁为本文的共同第一作者。北京合生基因科技有限公司刘兵亦对此文给予了一定的帮助。该成果得到了国家自然科学基金委重点仪器研发项目、面上项目,国家重点研发计划以及清华大学自主科研计划的资助。
来源:序说DNASpeaking
版权及免责声明:本网站所有文章除标明原创外,均来自网络。登载本文的目的为传播行业信息,内容仅供参考,如有侵权请联系答魔删除。文章版权归原作者及原出处所有。本网拥有对此声明的最终解释权。


{replyUser1} 回复 {replyUser2}:{content}