中国科学家合作解码人类基因组新型甲基化修饰
5mC是真核生物基因组DNA最主要的甲基化修饰方式,而6mA是原核生物基因组DNA最主要的甲基化修饰方式(6mA一般指DNA上的N6-甲基腺嘌呤,而更常见的m6A指代RNA上的修饰,二者要注意区分)。m6A甲基化修饰在真核生物RNA上频繁出现,其在RNA剪切、RNA稳定、RNA翻译和基因表达调控中扮演重要角色,目前已有大量的研究集中在相关领域【1,2】(Cell Research连续发表的三篇m6A相关综述,很多关键问题仍有待解决;特别推荐 | 凡是过去,皆为序章——m6A结构生物学前沿进展;激烈交锋丨Darnell父子与何川关于m6A修饰的“暗战”)。然而在真核生物中,基因组DNA的6mA甲基化修饰一度被认为不存在。随着深度测序技术的发展,当前在有限几种真核生物基因组DNA中发现6mA修饰的存在(衣藻、线虫、果蝇、真菌、爪蟾、拟南芥、猪、斑马鱼、四膜虫)【3-10】。
比较有争议的是,2016年耶鲁大学医学院Andrew Xiao课题组在Nature上报道了小鼠胚胎干细胞中的DNA上存在6mA修饰【11】,随后2017年德国的科学家在Angewandte Chemie International Edition杂志上反驳,声称用超高灵敏度质谱技术并没有在小鼠干细胞以及其它组织中检测到DNA的6mA修饰【12】。如果说DNA的6mA修饰在小鼠中尚存争议,那么人类基因组的6mA修饰岂不是更不可能了?(2016年华西的一项研究声称在人类间充质干细胞中发现DNA的6mA修饰对成骨分化有作用【13】)因此,目前关于人类基因组DNA上是否存在6mA修饰的问题一直没有得到解决。
7月12日,广州医科大学附属第三医院晏光荣团队、中山眼科中心肖传乐团队、美国费城儿童医院王凯教授、中国农业科学院生物技术研究所谷晓峰团队和军事科学院伯晓晨研究员合作在Molecular Cell 上,在线发表了题为N6-Methyladenine DNA Modification in Human Genome的研究论文,运用第三代测序中的PacBio单分子实时(Single Molecule Real-Time,SMRT)DNA测序方法,解码了人类基因组DNA中的6mA甲基化修饰,确定了调控人类基因组DNA 6mA甲基化修饰的甲基化酶N6AMT1和去甲基化酶ALKBH1,揭示了人基因组DNA 6mA水平下调,促使了肿瘤的发生发展和侵袭转移,并与肿瘤患者的预后差密切相关。

在这项研究中,研究人员采用SMRT测序技术对来自中国人的血基因组DNA进行深度测序(103 х coverage),鉴定发现人类基因组DNA中广泛存在6mA甲基化修饰(鉴定到了881240个6mA位点,占总的腺嘌呤约0.051%,这一丰度比此前报道的衣藻、线虫、真菌含量要低),线粒体基因组DNA上具有更高的6mA甲基化修饰(含量为0.184%,约为常染色体含量的三倍之多(下图));6mA修饰频繁出现在含有AGG motif的位点上,揭示6mA甲基化修饰显著富集在基因组外显子区,特别显著定位于基因转录终止位点,与基因的转录水平呈显著的正相关。

进一步研究发现,N6AMT1是调控人类基因组DNA 6mA修饰的甲基化酶(根据细菌中限制-修饰系统的DNA甲基转移酶推测然后验证),确定了N6AMT1上NPPY motif(122-125aa)是决定N6AMT1甲基化酶活性的关键结构域;发现ALKBH1是调控人类基因组DNA 6mA修饰的去甲基化酶(2016年Andrew Xiao课题组的结果显示ALKBH1是小鼠中的DNA 6mA去甲基化酶),并确定ALKBH1上233位的天冬氨酸(D233)是决定ALKBH1去甲基化酶活性的关键位点。
此外,该研究还进一步证实6mA和甲基化酶N6AMT1水平在人肿瘤组织中显著下调,并且N6AMT1基因在肿瘤组织中频繁缺失,而去甲基化酶ALKBH1水平在人肿瘤组织中显著上调。证实肿瘤组织中基因组DNA的6mA水平下调与肿瘤的恶性表型和肿瘤患者预后差密切相关。进而,揭示人基因组DNA 6mA水平的下调可能促使了肿瘤的发生发展和侵袭转移,而6mA水平的上调抑制了肿瘤的发生发展和侵袭转移。
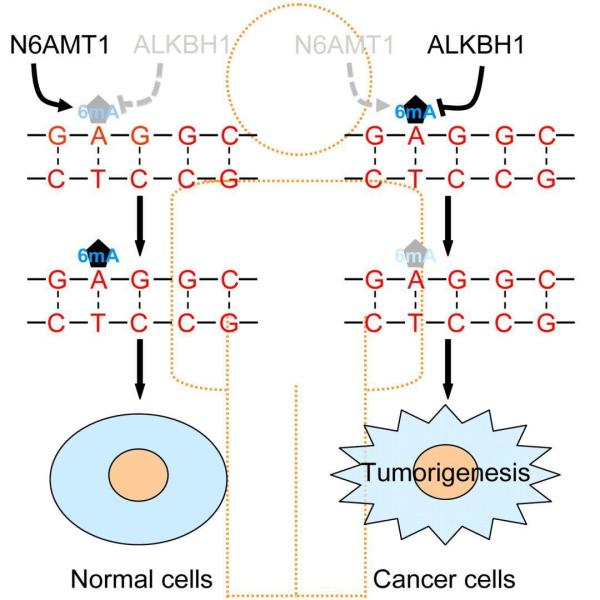
总的来说,该项研究工作首次证实了人类基因组DNA上6mA甲基化修饰的存在,并揭示了此6mA甲基化修饰的改变与肿瘤等疾病的发生发展有关,为肿瘤发病机制、新标志物和新干预靶点的选择提供了思路和依据,为今后深度解析基因组DNA 6mA甲基化修饰在胚胎发育、生理、病理和疾病中的功能角色奠定了基石。
应该说,本研究是系统研究人类基因组DNA 6mA修饰的开篇之作。然而,对于围绕人类基因组上DNA是否存在、存在多少6mA修饰的问题仍然需要更多课题组去验证,因为只有多个研究小组反复验证了,那么结论才能形成更广泛的共识。从生物学功能上讲,目前也需要更加细致的研究证据来确定DNA 6mA参与众多生物学过程调控。相关领域后续的研究报道仍然十分值得期待。
据悉,肖传乐、朱嵩、何铭辉是该论文并列第一作者。晏光荣研究员为该论文通讯作者,王凯教授、谷晓峰研究员和伯晓晨研究员是该论文共同通讯作者。
附第一作者和通讯作者简介
晏光荣研究员简介:

广州医科大学附属第三医院研究员、博士生导师、中心实验室/生物医学研究中心主任,珠江科技新星,羊城学者,广东省杰出青年卫生人才,广州高层次卫生重点医学人才。主要从事肿瘤发病机制研究。以第一或通讯作者在Mol Cell(2篇)、J Pathol等上发表SCI论文近30篇。获得教育部高校科研成果奖自然科学奖二等奖和湖南省自然科学奖一等奖等。
肖传乐副研究员简介:

中山眼科中心副研究员、广医三院客座教授,主要从事三代测序分析工具开发及应用研究,主持开发发表在Nature Methods上的三代测序组装系统MECAT。以第一和通讯作者在Nature Methods,Molecular Cell,NAR等杂志上发表高水平SCI论文二十余篇。先后开发MECAT,FANSe2和ProVerB等生物信息学分析工具。
王凯教授简介:

美国宾夕法尼亚大学病理系副教授,兼任费城儿童医院细胞与分子治疗中心研究员。发展了PennCNV, ANNOVAR, InterVar, RepeatHMM,Phenolyzer和EHR-Phenolyzer等基因组分析软件。发表第一个基于纯三代组装策略的亚洲人基因组接近完成图“华夏一号”。发表了150余篇文章,被引用25000余次。
谷晓峰研究员简介:

中国农业科学院生物技术研究所研究员、国家“青年千人”、中国农业科学院“青年英才计划”A类。主要从事表观遗传调控基因表达和发育机制的研究,近年以第一作者或通讯作者在Developmental Cell、PLoS Biology、Nature Communications、Molecular Plant等国际著名杂志发表多篇论文。
伯晓晨研究员简介:

军事科学院军事医学研究研究员、博士生导师,“万人计划”科技领军人才,中国科协“求是”杰出青年奖获得者。在Nature、Nature Microbiology、Molecular Cell等期刊发表SCI论文70余篇;先后主持“精准医学研究”国家重点研发专项、国家重大新药创制、863、自然基金重大研究计划等项目18项。
论文原文:N6-Methyladenine DNA Modification in the Human Genome
参考文献:
1. Coots RA, Liu XM, Mao Y, et al. m(6)A Facilitates eIF4F-Independent mRNA Translation. Mol Cell. 2017, 68(3):504-514.
2. Xiao W, Adhikari S, Dahal U, et al. Nuclear m(6)A Reader YTHDC1 Regulates mRNA Splicing. Mol Cell.2016, 61(4):507-519.
3. Fu Y, Luo GZ, Chen K,et al. N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas. Cell. 2015, 161(4):879-892.
4. Zhang G, Huang H, Liu D, et al.N6-methyladenine DNA modification in Drosophila. Cell. 2015, 161(4):893-906.
5. Greer EL, Blanco MA, Gu L,et al. DNA Methylation on N6-Adenine in C. elegans. Cell. 2015, 161(4):868-78.
6. Mondo SJ, Dannebaum RO, Kuo RC, et al. Widespread adenine N6-methylation of active genes in fungi. Nat Genet. 2017, 49(6):964-968.
7. Koziol, M. J., Bradshaw, C. R., Allen, G. E., Costa, A. S., Frezza, C., & Gurdon, J. B. (2016). Identification of methylated deoxyadenosines in vertebrates reveals diversity in DNA modifications. Nature structural & molecular biology, 23(1), 24.
8. Liang, Z., Shen, L., Cui, X., Bao, S., Geng, Y., Yu, G., ... & Yu, H. (2018). DNA N6-Adenine Methylation in Arabidopsis thaliana. Developmental cell, 45(3), 406-416.
9. Liu, J., Zhu, Y., Luo, G. Z., Wang, X., Yue, Y., Wang, X., ... & Han, D. (2016). Abundant DNA 6mA methylation during early embryogenesis of zebrafish and pig. Nature communications, 7, 13052.
10. Wang, Y., Sheng, Y., Liu, Y., Pan, B., Huang, J., Warren, A., & Gao, S. (2017). N6-methyladenine DNA modification in the unicellular eukaryotic organism Tetrahymena thermophila. European journal of protistology, 58, 94-102.
11. Wu, T. P., Wang, T., Seetin, M. G., Lai, Y., Zhu, S., Lin, K., ... & Tackett, A. (2016). DNA methylation on N 6-adenine in mammalian embryonic stem cells. Nature, 532(7599), 329.
12. Schiffers, S., Ebert, C., Rahimoff, R., Kosmatchev, O., Steinbacher, J., Bohne, A. V., ... & Carell, T. (2017). Quantitative LC–MS provides no evidence for m6dA or m4dC in the genome of mouse embryonic stem cells and tissues. Angewandte Chemie International Edition, 56(37), 11268-11271.
13. Zhou, C., Liu, Y., Li, X., Zou, J., & Zou, S. (2016). DNA N 6-methyladenine demethylase ALKBH1 enhances osteogenic differentiation of human MSCs. Bone research, 4, boneres201633.
来源:BioArt
版权及免责声明:本网站所有文章除标明原创外,均来自网络。登载本文的目的为传播行业信息,内容仅供参考,如有侵权请联系答魔删除。文章版权归原作者及原出处所有。本网拥有对此声明的最终解释权。


{replyUser1} 回复 {replyUser2}:{content}