500名科学家倾力合作,地球微生物组计划首发成果
地球不仅是人类生活的家园,也是各种微生物栖息的场所。不过,微生物的数量如此庞大,要想给它们列个清单,那可并非易事。近日,一个国际研究联盟在《Nature》杂志上公布了地球微生物组计划(Earth Microbiome Project)的首个成果,成功迈出了第一步。

专家估计,地球上大约有一万亿种微生物。事实上,海洋中的细菌比宇宙中的星星还要多。这些细菌几乎无处不在:人类肠道、亚马逊丛林、火山喷发口、大西洋海底……不过,基于种种原因,全面鉴定这些微生物可是一项极具挑战性的任务。
地球微生物组计划于2010年启动,最终目标是鉴定世界各地的20万个微生物样本,以生成一份详细的目录。如今,这个联盟已经从全球科学界收集了27,000多个样本。利用这些样本,研究人员鉴定出大约30万条独特的微生物16S rRNA序列,其中绝大多数无法在现有数据库中找到。
这项工作体现了全球生物界的通力合作。论文第一作者、加州大学圣地亚哥分校的Luke Thompson表示:“40多个国家的500多名研究人员向我们提供了样本和数据。我们不仅有饮用水和漏油相关的样本,还有各种动物的皮肤样本,包括海豚、海豹、鱼、鸟、蝙蝠、响尾蛇等。这个项目的协作真的很激动人心。”
 Rob Knight
Rob Knight
加州大学圣地亚哥分校的Rob Knight表示:“这个数据库的潜在应用几乎是无限的。举个例子,我们现在可以通过分析微生物组,或分析微生物的类型和相对数量,确定样本来自何种类型的环境。这也许能帮助法医获得犯罪现场的信息。想想CSI吧。”
 地球微生物组计划图
地球微生物组计划图
在这项研究中,研究人员重点关注了首批27,751个样本的细菌和古细菌及其元数据。研究人员打造了一个轻量级的应用程序,以确定样本是自由生活的,还是依附于宿主,如果是后者,那么宿主是植物还是动物。它还确定了样本是来自盐水还是非盐水环境。
研究人员对这些样本的16S rRNA基因进行扩增,并在Illumina平台上开展测序。他们总共产生了22亿条序列。Knight指出,研究人员对之前的许多操作方案进行了测试。他们倾向于普遍适用的方案,而非灵敏度高的方案。
与以往的许多宏基因组研究不同,Knight及其同事并不是将序列分配到操作分类单元(OTU),而是利用一种最近开发的无参考方法来获取确切的序列。在质量控制之后,他们生成了307,572条独特序列。在这些序列中,只有10%与现有的数据库相匹配。这种方法让研究人员能够追踪样本中的rRNA基因序列,并探索环境的微生物多样性。
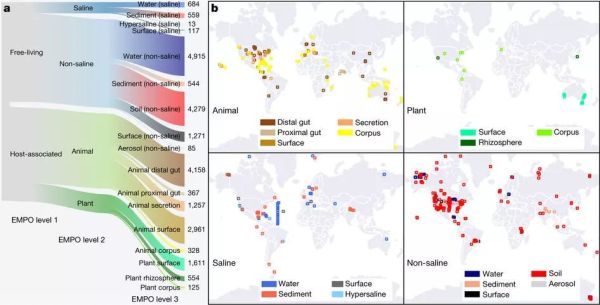
研究人员证实了以前的发现,依附于宿主意味着丰富度降低,而盐水和非盐水环境有着组成不同的微生物群落。例如,鲸类动物(鲸和海豚)及鱼类的皮肤微生物组与它们生活的水域相似。不过,盐水使它们的微生物组与淡水中的生物不同。
此外,一种机器学习方法可以根据群落组成来区分样本,包括是否与动植物相关联,是否来源于盐水环境,准确度达到91%。他们认为,这将有助于法医及其他应用。
此外,研究人员还报告称,正如预期的那样,在中性pH和相对较冷的环境中的微生物丰富度是最高的。他们还发现,嵌套性(nestedness)似乎在这些群落中占主导地位,这意味着多样性低的群落可能是多样性高的群落的子集,而不是完全不同的群体。
另一位通讯作者、西北太平洋国家实验室的Janet Jansson表示:“我们还需要更多的采样来考虑纬度和海拔等因素,并追踪环境随时间而发生的改变。地球微生物组计划为探索无数问题提供了资源,也成为解答各种问题的起点。”


{replyUser1} 回复 {replyUser2}:{content}