Cell:施一公团队解析酵母ILS状态剪接体
北京时间11月17日凌晨,来自清华大学生科院,西湖大学的研究人员在Cell在线发表了题为“Structure of an Intron Lariat Spliceosome from Saccharomyces cerevisiae”的文章,报道了RNA剪接循环中剪接体最后一个状态的高分辨率三维结构,为阐明剪接体完成催化功能后受控解聚的分子机制提供了结构基础,从而将对RNA剪接(RNA Splicing)分子机理的理解又推进了一步。
 最近,Nature还制作了一个mRNA剪接40周年专题页面(下图),该专题将Nature系列杂志中的部分近期发表的代表性论文都精选了出来。
最近,Nature还制作了一个mRNA剪接40周年专题页面(下图),该专题将Nature系列杂志中的部分近期发表的代表性论文都精选了出来。
 值得一提的是,施一公教授撰写的题为“Mechanistic insights into precursor messenger RNA splicing by the spliceosome”的综述发表在Nature Reviews Molecular Cell Biology杂志上,而英国MRC的Kiyoshi Nagai等撰写的题为“Cryo-electron microscopy snapshots of the spliceosome: structural insights into a dynamic ribonucleoprotein machine”的综述则是发表在Nature Structural & Molecular Biology杂志上,两篇综述文章差不多在同一期间发表。另外,来自耶鲁大学的 Karla M. Neugebauer等人也差不多稍早前在Nature Reviews Molecular Cell Biology杂志上发表了题为“Splicing and transcription touch base: co-transcriptional spliceosome assembly and function”的综述(下图)。
值得一提的是,施一公教授撰写的题为“Mechanistic insights into precursor messenger RNA splicing by the spliceosome”的综述发表在Nature Reviews Molecular Cell Biology杂志上,而英国MRC的Kiyoshi Nagai等撰写的题为“Cryo-electron microscopy snapshots of the spliceosome: structural insights into a dynamic ribonucleoprotein machine”的综述则是发表在Nature Structural & Molecular Biology杂志上,两篇综述文章差不多在同一期间发表。另外,来自耶鲁大学的 Karla M. Neugebauer等人也差不多稍早前在Nature Reviews Molecular Cell Biology杂志上发表了题为“Splicing and transcription touch base: co-transcriptional spliceosome assembly and function”的综述(下图)。
 据报道,真核生物的基因表达相比于原核生物,更为复杂也更为精细。由于真核细胞 内的基因是不连续的,它需要在细胞核内被转录成前体信使 RNA,通过RNA 剪接,不具有翻译功能的内含子被去除,密码子所在的外显子被连接,从而得到成熟的、可被翻译成蛋白质的信使 RNA。 RNA 剪接是真核生物基因表达调控的重要环节之一,而负责执行这一过程的是细胞核内一个巨大的且高度动态变化的分子机器——剪接体(spliceosome)。剪接体在真核生物进化中极为保守,对于真核生物维持正常的生命活动至关重要。一个基因转录出的前体信使RNA 可以通过RNA剪接成若干种信使RNA,于是极大地丰富了真核生物蛋白质组的多样性。在剪接反应过程中,多种蛋白质-核酸复合物及剪接因子按照高度精确的顺序发生结合和解聚,依次形成预组装复合物U4/U6.U5 Tri-snRNP以及至少7个状态的剪接体B、Bact、B*、C、C*、P以及ILS复合物(下图)。
据报道,真核生物的基因表达相比于原核生物,更为复杂也更为精细。由于真核细胞 内的基因是不连续的,它需要在细胞核内被转录成前体信使 RNA,通过RNA 剪接,不具有翻译功能的内含子被去除,密码子所在的外显子被连接,从而得到成熟的、可被翻译成蛋白质的信使 RNA。 RNA 剪接是真核生物基因表达调控的重要环节之一,而负责执行这一过程的是细胞核内一个巨大的且高度动态变化的分子机器——剪接体(spliceosome)。剪接体在真核生物进化中极为保守,对于真核生物维持正常的生命活动至关重要。一个基因转录出的前体信使RNA 可以通过RNA剪接成若干种信使RNA,于是极大地丰富了真核生物蛋白质组的多样性。在剪接反应过程中,多种蛋白质-核酸复合物及剪接因子按照高度精确的顺序发生结合和解聚,依次形成预组装复合物U4/U6.U5 Tri-snRNP以及至少7个状态的剪接体B、Bact、B*、C、C*、P以及ILS复合物(下图)。
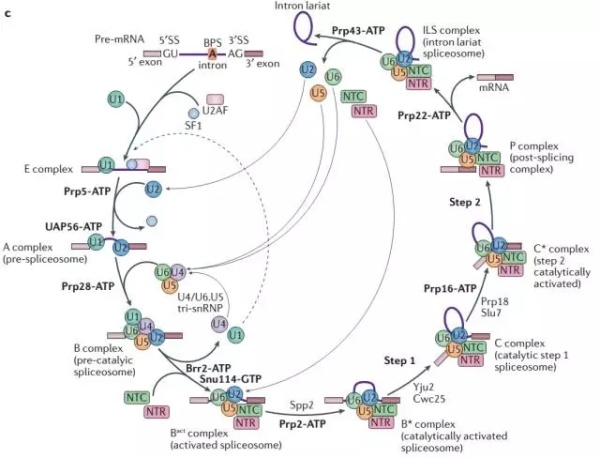 RNA剪接示意图(图片来源: Shi Y. Journal of Molecular Biology, 2017)
RNA剪接示意图(图片来源: Shi Y. Journal of Molecular Biology, 2017)
在经过近四十年的遗传和生化研究之后,剪接体介导的pre-mRNA的剪接过程的神秘的面纱逐渐被揭开。早在2015年8月,施一公课题组就在Science上背靠背地发表两篇研究长文,报道了裂殖酵母3.6 Å分辨率下的内含子套索剪切体ILS complex的冷冻电镜结构,揭示了剪接体剪接活性位点的原子详情和总的蛋白和RNA的组织形式,并阐释了剪切体对pre-mRNA进行剪接的基本工作原理。此后,施一公教授课题组与MRC分子生物学实验室Nagai研究组、德国马普生物物理化学研究所Reinhard Lüehrmann教授一起互补或背靠背地解析了酿酒酵母或人源的预组装复合物U4/U6.U5 tri-snRNP、催化前复合物B complex、激活状态复合物Bact complex、第一步催化反应复合物C complex、第二步催化激活状态下的C* complex和内含子套索剪接体ILS complex。除了B complex,其它四个状态下的主要的结构特色都与裂殖酵母ILS complex的一致。这样,就只剩下催化激活的复合物B* complex和催化后复合物P complex在结构上未被表征。
在新解析的酿酒酵母3.6 Å的P complex的冷冻结构电镜中,对36个剪接体蛋白,3个snRNA(U2、U5和U6),1个连接的外显子和1个内含子套索进行了原子建模。最终的结构模型包括了9328个氨基酸酸残基和381个RNA核苷酸,联合起来的分子质量达到了~1.2 MD。如之前预测的一样,P complex的总的组织形式和详细的结构特色与C* complex的非常类似。
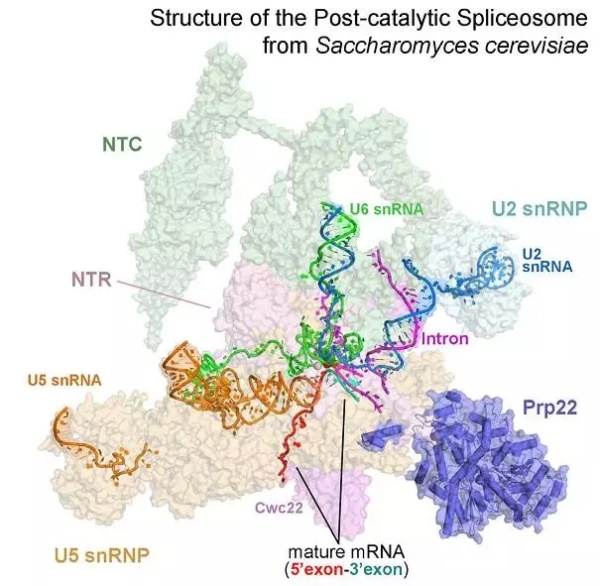 在P complex中,步骤II剪接因子Prp17, Prp18和Slu7仍结合在活性位点附近。剪接因子Cwc21和Cwc22一起稳定了外显子。ATP酶/解螺旋酶Prp22位于高度不对称的P complex的一个角落,主要与Prp8的Lingker结构域相互作用。连接的外显子锚定在U5 snRNA的loop上,保守的3’-拼接位点(3 ‘SS)的AG二核苷酸则坐落于活性位点。
在P complex中,步骤II剪接因子Prp17, Prp18和Slu7仍结合在活性位点附近。剪接因子Cwc21和Cwc22一起稳定了外显子。ATP酶/解螺旋酶Prp22位于高度不对称的P complex的一个角落,主要与Prp8的Lingker结构域相互作用。连接的外显子锚定在U5 snRNA的loop上,保守的3’-拼接位点(3 ‘SS)的AG二核苷酸则坐落于活性位点。
尽管P complex的结构高度类似于C* complex,该结构仍然在剪接体的活性位点处揭示了意想不到的发现,该发现对我们机制的理解pre-mRNA的拼接具有十分重要的启示作用。同时结构的阐释使得我们理解了剪切体C*到P 和 the P到ILS 的转变机制,因而是完成pre-mRNA剪接循环的必须一步。我们有理由期待施一公团队在B* complex的结构上的再次领先。所有状态下剪接体(近)原子分辨率下的结构解析,将对剪接体的组装、激活、催化和分解过程提供完整的机制上的见解。
据悉,清华大学施一公教授研究组一直致力于捕捉RNA剪接过程中处于不同动态变化的剪接体结构,从而从分子层面阐释RNA剪接的工作机理。2015年,施一公研究组率先突破,在世界上首次报道了裂殖酵母剪接体3.6埃的高分辨率结构,首次展示了剪接体催化中心近原子分辨率的结构。自2015年第一个剪接体结构发表以后,施一公研究组相继解析了5个不同状态剪接体复合物的高分辨率结构,分别是酿酒酵母3.8埃的预组装复合物U4/U6.U5 Tri-snRNP、3.5埃的激活状态复合物Bact complex、3.4埃的第一步催化反应后复合物C complex、4.0埃的第二步催化激活状态下的C* complex,以及本文3.5埃的内含子套索剪接体ILS complex的结构。这个5个不同状态的剪接体基本覆盖了整个剪接通路中从预组装到激活、从发生两步转酯反应到剪接体的解聚的关键催化步骤,呈现了迄今为止最为清晰的剪接体不同工作状态下的结构信息,将RNA剪接领域的发展推向了新的高度。施一公教授因此于不久前刚刚获得未来科学大奖生命医学奖。
参考文献:
1.Shi, Y. (2017). Mechanistic insights into precursor messenger RNA splicing by the spliceosome. Nature Reviews Molecular Cell Biology.
2、Fica, S. M., & Nagai, K. (2017). Cryo-electron microscopy snapshots of the spliceosome: structural insights into a dynamic ribonucleoprotein machine. nature structural & molecular biology, 24(10), 791-799.
3、Herzel, L., Ottoz, D. S., Alpert, T., & Neugebauer, K. M. (2017). Splicing and transcription touch base: co-transcriptional spliceosome assembly and function. Nature Reviews Molecular Cell Biology.


{replyUser1} 回复 {replyUser2}:{content}