|
2025-01-27 07:18:22
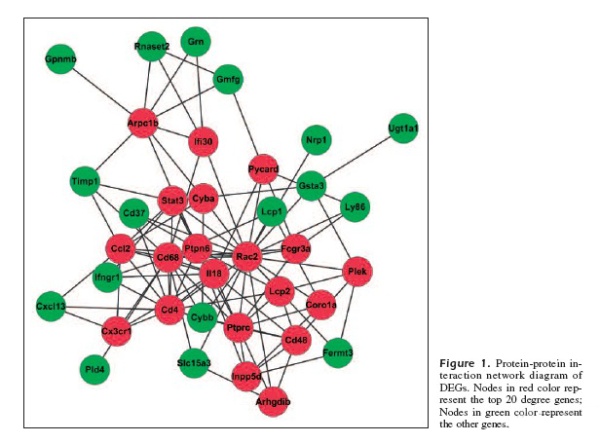
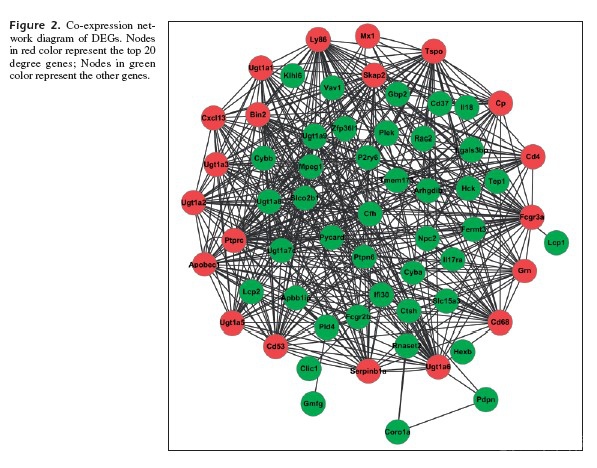
coexpression network是如何做出来的?最近在学习文献的时候发现coexpression network,这个共表达网络和cytoscape做出来的PPI network有什么区别呢?在Identification crucial genes in peripheral neuropathic pain induced by spared nerve injury 这篇文章里作者把coexpression network和PPI network都做了,如下图: coexpression network:
|
2个回答
|
这是很初级的网络重构。 PPI network通常是从蛋白质互作数据库,比如HPRD/DIP等,提取全部或感兴趣的互作蛋白质对,然后使用网络可视化工具,如Cytoscape等,绘制网络图。共表达网络,通常是通过计算基因之间的相似性测度,比如pearson correlation coefficient,选取一定的阈值,从而得到共表达基因对,并以此构建网络。可以说,PPI network是实验测定的网络,属于生物网络可视化;基因共表达网络是尝试通过基因之间的表达一致性关系,重构生物学网络,期望发现新规律。类似的网络还有,转录因子调控网络,microRNA调控网络,疾病关系网络等等。Cytoscape最早只是个网络可视化和网络分析工具,随着它越来越多的应用于生物网络构建/重构,研究人员开发了各种不同的插件,极大的简化了网络构建的步骤。值得注意的是,研究人员需要正确运用各种插件,理解插件所能提供的信息,根据具体的分析需求选用,而不是随便弄一套数据,应用一个插件,构建出你自己都不知为何物的“网络”。 |
你的回答
| 提问时间: | 2025-01-27 |
| 浏览量: | 2538 |
| 最近回答: | 2025-01-27 |